最近對16s的三大資料庫的序列的具體序列情況挺好奇的,決定統計一下各個序列的長度分布情況,以及這些序列具體分布在哪幾個V區,有助于我解決後面16So資料的問題。還是用上我三腳貓的功夫,開始今天 的探索,沒有人探索的事情,還是挺開心的。
1.統計序列長度分布情況
01
#獲得長度清單檔案
02
length_list = []
03
with open('current_Bacteria_unaligned.fa') as f:
04
flag = 0
05
length = 0
06
for line in f:
07
if line.startswith('>'):
08
flag = 1
09
if length != 0:
10
#print(length)
11
length_list.append(length)
12
#break
13
length = 0
14
else:
15
length = 0
16
elif flag == 1:
17
length +=len(line)
18
19
fout = open('length_table.txt', 'w')
20
for a in length_list:
21
fout.write(str(a) + '\n')
22
fout.close()
01
#統計長度區間分布
02
length_150 = 0
03
length_300 = 0
04
length_600 = 0
05
length_1300 = 0
06
length_1300_ = 0
07
with open('length_table.txt') as f:
08
for line in f:
09
if 0 < int(line.strip()) <= 150:
10
length_150 += 1
11
elif 150 < int(line.strip()) <= 300:
12
length_300 += 1
13
elif 300 < int(line.strip()) <= 600:
14
length_600 += 1
15
elif 600 < int(line.strip()) <= 1300:
16
length_1300 += 1
17
elif int(line.strip()) > 1300:
18
length_1300_ += 1
19
print('150以内:', length_150,'\n',
20
'150-300:', length_300, '\n',
21
'300-600:', length_600, '\n',
22
'600-1300:', length_1300,'\n',
23
'1300-:', length_1300_)
1
#最後結果
2
150以内: 0
3
150-300: 0
4
300-600: 617,554
5
600-1300: 1,063,377
6
1300-: 1,515,109 複制
我也可視化一下:
更正一下,這裡用的是RDP資料庫,之前由于Silva資料庫用的是不相容的14級分類系統而沒采用。
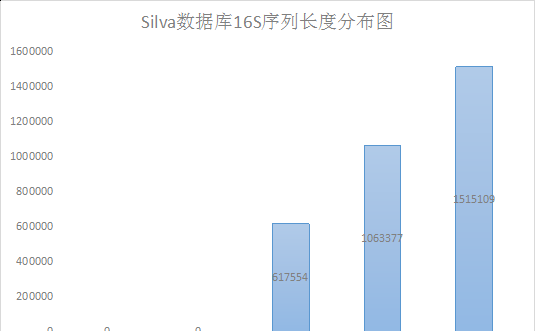
從圖中可以看出,大部分序列還是集中在600以上。
接着是greengenes資料庫,這個資料庫雖然序列較少,但是長度大部分集中在1300+,品質較高,就是好久沒更新過了。
150以内: 0
150-300: 0
300-600: 0
600-1300: 895
1300-: 1262090
2.統計V區分布情況
從一個公衆号得到的一張分布圖是這樣的,
我想确定的是序列都包含在哪個或者哪兩個區。花了好大有力氣,才把代碼理好,效率應該還是比較低的,但是,對于我來說
001
import re
002
003
dic ={}
004
v1f = re.compile('GGATCCAGACTTTGATYMTGGCTCAG', re.I)
005
v3f = re.compile('CCTA[CT]GGG[AG][GTC]GCA[CG]CAG', re.I)
006
v4f = re.compile('GTG[CT]CAGC[AC]GCCGCGGTAA', re.I)
007
v4r = re.compile('ATTAGA[AT]ACCC[CTG][ATGC]GTAGTCC', re.I)
008
v6f = re.compile('AACGCGAAGAACCTTAC', re.I)
009
v8f = re.compile('CGTCATCC[AC]CACCTTCCTC', re.I)
010
vr = re.compile('AAGTCGTAACAAGGTA[AG]CCGTA', re.I)
011
#v4r = re.compile('GGACTAC[ATGC][ACG]GGGT[AT]TCTAAT', re.I)
012
vf = re.compile('AG[AG]GTT[CT]GAT[CT][AC]TGGCTCAG', re.I)
013
#vr = re.compile('GACGGGCGGTG[AT]GT[AG]CA', re.I)
014
#seq_list = []
015
016
017
def decide_which_zone(f1, f3, f4, f4r, f6, f8, f, fr):
018
type = [f1, f3, f4, f4r, f6, f8, f, fr]
019
type_str = ['f1', 'f3', 'f4', 'f4r', 'f6', 'f8', 'f', 'fr']
020
type_name = []
021
for i in range(len(type)):
022
#print(type[i])
023
if type[i] != None :
024
type_name.append(type_str[i])
025
else:
026
continue
027
if i + 1 < len(type):
028
if type[i + 1] != None and type_str[i + 1] not in type_str:
029
type_name.append(type_str[i + 1])
030
else:
031
continue
032
if i + 2 < len(type):
033
if type[i + 2] != None and type_str[i + 2] not in type_str:
034
type_name.append(type_str[i + 2])
035
else:
036
continue
037
if i + 3 < len(type):
038
if type[i + 3] != None and type_str[i + 3] not in type_str:
039
type_name.append(type_str[i + 3])
040
else:
041
continue
042
if i + 4 < len(type):
043
if type[i + 4] != None and type_str[i + 4] not in type_str:
044
type_name.append(type_str[i + 4])
045
else:
046
continue
047
if i + 5 < len(type):
048
if type[i + 5] != None and type_str[i +5 ] not in type_str:
049
type_name.append(type_str[i + 5])
050
else:
051
continue
052
if i + 6 < len(type):
053
if type[i + 6] != None and type_str[i + 6] not in type_str:
054
type_name.append(type_str[i + 6])
055
else:
056
continue
057
if i + 7 < len(type):
058
if type[i + 7] != None and type_str[i + 7] not in type_str:
059
type_name.append(type_str[i + 7])
060
else:
061
continue
062
return type_name
063
064
with open('current_Bacteria_unaligned.fa') as f:
065
li = ['f1', 'f3', 'f4', 'f4r', 'f6', 'f8', 'f', 'fr']
066
for i in range(len(li)):
067
for j in range(len(li)):
068
if i <= j:
069
dic[li[i] + li[j]] = 0
070
071
072
#print(dic)
073
flag = 0
074
seq = ''
075
#i = 0
076
for line in f:
077
if line.startswith('>'):# and i <=2:
078
flag = 1
079
if seq != '':
080
#print(seq)
081
f1 = v1f.search(seq)
082
f4r = v4r.search(seq)
083
f3 = v3f.search(seq)
084
f4 = v4f.search(seq)
085
f6 = v6f.search(seq)
086
f8 = v8f.search(seq)
087
f = vf.search(seq)
088
fr = vr.search(seq)
089
#seq_list.append(length)
090
type_name = []
091
type_name = decide_which_zone(f1, f3, f4, f4r, f6, f8, f, fr)
092
print(type_name)
093
if type_name != []:
094
type_key = type_name[0] + type_name[-1]
095
dic[type_key] += 1
096
#i += 1
097
#break
098
seq = ''
099
flag = 0
100
else:
101
seq = ''
102
elif flag == 1:
103
seq += line.strip()
104
print(dic)
105
#運作結果
106
{'f1f1': 0, 'f1f3': 0, 'f1f4': 1, 'f1f4r': 0, 'f1f6': 0, 'f1f8': 0, 'f1f': 0, 'f1fr': 0, 'f3f3': 120867, 'f3f4': 143686, 'f3f4r': 356027, 'f3f6': 400108, 'f3f8': 4, 'f3f': 171637, 'f3fr': 53716, 'f4f4': 17211, 'f4f4r': 71719, 'f4f6': 40223, 'f4f8': 4, 'f4f': 13185, 'f4fr': 3646, 'f4rf4r': 39537, 'f4rf6': 45160, 'f4rf8': 0, 'f4rf': 977, 'f4rfr': 4263, 'f6f6': 43983, 'f6f8': 0, 'f6f': 64, 'f6fr': 2300, 'f8f8': 0, 'f8f': 0, 'f8fr': 0, 'ff': 1670, 'ffr': 26, 'frfr': 3678} 複制
上圖依然是RDP資料庫,前面标錯了。應該是某個地方出了問題,序列不全,少了估計有一半。不過趨勢還是比較明顯的,V3-V4是最多的,這也是前些年主流的研究的測序方式。