發現一個中國疾控中心的從業人員zhangwen2001做的一個docker,剛好最近在分析腸道微生物的資料,學習學習。動用我的自以為比較厲害的搜尋和查找資訊的能力,終于找到了鏡像檔案。先把github的位址放在這:
https://github.com/zhangwen2001/Guthealthy
這個代碼庫并沒有說明docker hub中有這個鏡像,但是我懷着僥幸心理搜尋了一下,是可以找到的,開心呀!作者的郵箱是[email protected],試了一下,icdc.cn網站是中國疾控的,是以我判斷作者是這個機關的人。
一、鏡像擷取
不過pull這個鏡像的過程中還是遇到了一點小問題,提示沒有預設的latest版本,于是我嘗試使用docker hub上标明的v1.0,成功地拉取了下來,當然,docker安裝也是有點小費勁,如果有報錯的話,還好有官方教程在。
docker pull zhangwen2001/guthealthy:v1.0 複制
二、鏡像使用
sudo docker run -it --rm -v /home/zd/microbiome_gut:/test zhangwen2001/guthealthy:v1.0 複制
cd /test 複制
perl /bioapp/bin/Guthealthy.pl /test/input.fq /test 複制
得到的報告大概就和軟體倉庫上介紹的差不多,可以獲得的資訊有:
1.彙總資訊
包括總的reads數,有多少個屬,香農指數
2.核心菌群資訊
各個菌屬的含量,以及正常人的菌群含量平均值
3.緻病菌的情況
總共分為三組,從未在正常人中發現的菌;從未在HMP計劃健康人中發現的菌;以及其他可能的緻病菌。
三、鏡像流程初步學習
從運作指令來看,這是一個perl腳本,打開來看了一下發現基本上是把各個軟體的串聯起來,對于我這個perl門外漢也能看懂,竊喜。作者的代碼已經注釋的相當清楚,第一步,質控,運作了自己的腳本去過濾低品質reads,輸出檔案裡還有一個fastqc的結果,從序列品質圖來看,質控做得相當漂亮,有圖為證。
質控之前的品質分布圖:
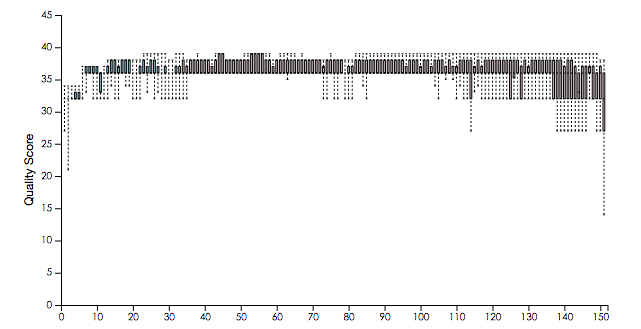
質控之後的序列品質分布圖:
第二步,統計細菌的多樣性,作者沒有使用最廣泛使用的qiime、mothur、vsearch等軟體,而是使用了bowtie2去比對一個自建的參考序列集。然後後面同樣用bowtie2獲得了緻病菌的相關資訊。話說我也曾經想過用比對去解決雙向測序長度短序列不能較好拼接的問題,苦于沒有思路,作者的思路值得我深入學習。最後一步是用一個cat指令生成了報告。
#!/usr/bin/perl 複制
use strict; 複制
use warnings; 複制
my $file=$ARGV[0]; #fq檔案 複制
my $out=$ARGV[1]; 複制
print "Step 1: Quality Control for Sequence Data 測序資料品質控制\n"; 複制
system "perl /bioapp/bin/read_filter_QC.pl -input $file -outdir $out\n"; 複制
print "Step 2: Genus Num 樣本中菌屬多樣性\n"; 複制
system "/bioapp/bin/bowtie2-2.3.2/bowtie2 -x /bioapp/Data/16S-complete-clean.fa -q $out/filter.fq -S filter.sam\n"; 複制
system "perl /bioapp/bin/bowtie_stat-v3.pl filter.sam /bioapp/Data/16S-complete-clean.taxon $out/filter.fq /bioapp/Data/Core_genus filter.stat\n"; 複制
print "Step 3:Pathogen Detection 樣本中含有的病原菌\n"; 複制
system "/bioapp/bin/bowtie2-2.3.2/bowtie2 -x /bioapp/Data/155pathogens.fa -q $out/filter.fq -S filter.bwa.sam\n"; 複制
#system "/bioapp/bin/bwa-0.7.12/bwa mem /bioapp/Data/155pathogens.fa $out/filter.fq >filter.bwa.sam\n"; 複制
system "perl /bioapp/bin/bwa_stat-v2.pl filter.bwa.sam /bioapp/Data/16S-complete-clean.taxon /bioapp/Data/pathogen.group filter.bwa.sam.stat\n"; 複制
print "Step 4: Report 報告生成\n"; 複制
system "cat filter.stat.genus filter.bwa.sam.stat.species >Report.txt\n"; 複制