差異基因篩選
差異系數,可以展現對象資料與标準資料的相對差異,數值越大,表示不平衡程度越大[1]
設x與y分别為對象資料和标準資料,則差異系數k的表達式為:
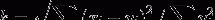
差異倍數
差異表達基因分析:差異倍數(fold change), 差異的顯著性(P-value) | 火山圖 - Life·Intelligence - 部落格園Differential gene expression analysis:差異表達基因分析 Differentially expressed gene (DEG):差異表達基因 Volcano Pl
關于R中p值的了解_yangnuanyang的部落格-CSDN部落格_r語言p值進行線性回歸lm後執行summary函數之後,會有Coefficients:Estimate Std. Error t value Pr(>|t|) 這樣的值出現,其中P值我是這樣了解的P值是用來判定假設檢驗結果的一個參數,也可以根據不同的分布使用分布的拒絕域進行比較。P值(P value)就是當原假設為真時所得到的樣本觀察結果或更極端結果出現的機率。這樣定義原假設,我們希望成立實作的研究假...javascript:void(0)
df = read.delim('DEG_nofiltered.xls',
header = T, sep = '\t')
#### 3.1.1 根據差異倍數大于2且P值小于0.05篩選
dim(subset(df, FC > 2 & pval < 0.05 |
FC < 0.5 & pval < 0.05))
df$logFC = log2(df$FC)
deg = subset(df, abs(logFC) > 1 & pval < 0.05)
dim(deg) ### 3.2 排序
#### 3.2.1 差異最顯著的50個基因
orderP = order(deg$pval) # 傳回pval從小到大排序的坐标
deg_orderP = deg[orderP,] # 根據上述位置關系重新排列行
top50P = head(deg_orderP, 50) # 提取前50行
top50P$id #### 3.2.2 差異倍數最大的50個基因
orderFC = order(abs(deg$logFC),
decreasing = T) # 傳回差異倍數由大到小排序的坐标
deg_orderFC = deg[orderFC,]
top50FC = head(deg_orderFC, 50)
top50FC$id 将資料寫入
### 4.2 txt檔案
write.table(deg_TF_all, 'deg_TF_all.xls',
col.names = T, row.names = F,
sep = '\t', quote = F)
### 4.3 xlsx檔案
library(writexl)
write_xlsx(list(mysheet=deg_TF_all),
'deg_TF_all.xlsx')