大家好,又見面了,我是你們的朋友全棧君。
【基因融合的定義】
基因融合(gene fusion)是指由于某種機制(如基因組變異)使得兩個不同基因的部分序列或全部序列融合到一起,形成了一個新的基因。如下圖所示:
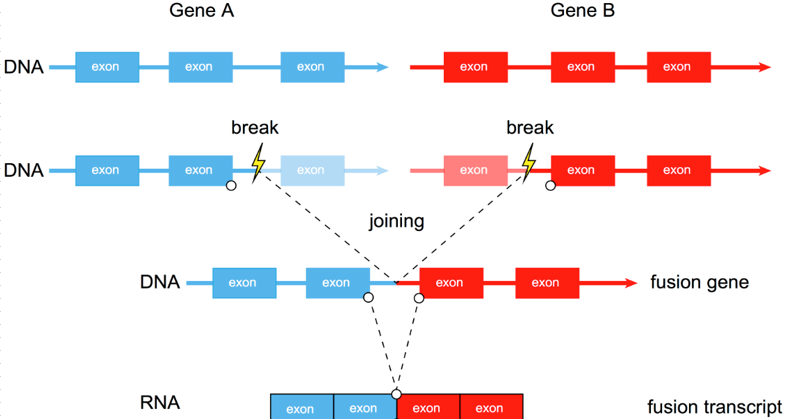
基因融合示意圖(圖檔來源:https://www.tumorfusions.org/)
一般來說,基因融合是指基因組層面的融合。但轉錄組層面也可能發生融合,主要是由于兩個不同基因轉錄産生的RNA,由于某種原因融合在了一起,形成新的融合RNA,該RNA可能編碼蛋白,也可能為非編碼。而基因組層面産生的融合基因,根據融合的情況,可能表達,也可能不表達(如破壞了啟動子區域或其他原因)。
【基因融合的産生機制】
基因融合的産生機制主要有以下三種,具體如下圖所示:
基因融合常見的三種發生機制(圖檔來源:Wikipedia)
基因融合常見的三種發生機制:
1)Chromosomal Translocation,染色體易位。如上圖A中1号和2号染色體上的兩片段發生交叉互換,導緻1号染色體上的淺綠色基因與2号染色體上的橘黃色基因融合到一起;
2)Interstitial deletion,中間缺失。如上圖中,3号染色體上的橘黃色基因和淺綠色基因之間的區段發生缺失(deletion),最終導緻這兩個基因融合到了一起;
3)Chromosomal Inversion,染色體倒位。如4号染色體上的橘黃色基因到墨綠色基因之間的片段發生倒位,最終導緻橘黃色基因和淺綠色基因融合到了一起。
【基因融合與癌症的關系】
那為什麼要研究基因融合呢?因為過去的諸多研究不斷表明,基因融合與各種疾病,特别是癌症的發生發展緊密相關,甚至是一些癌症的直接誘因,是以基因融合也成為了目前組學大資料分析中的一項重要研究内容。
目前,已經報道有很多癌症的發生與基因融合密切相關,具體如下表所示:
已知的一些惡性良性腫瘤中的基因融合數量和複發融合數量 (圖檔來源:Mertens et al. Nature Reviews Cancer, 2015)
而且,美國FDA(Food and Drug Administration)已經準許了一些針對特定基因融合的藥物,以治療相應的癌症,具體如下表所示:
FDA已準許的治療相應惡性良性腫瘤的藥物 ( 圖檔來源:Mertens et al. Nature Reviews Cancer, 2015)
是以,基因融合可能與各種癌症的發生發展緊密相關,這些融合基因還可能是潛在的藥物靶點,非常有必要對它們進行深入的研究。
【基于全基因組測序和轉錄組測序鑒定基因融合】
基因融合的鑒定,可以基于全基因組測序資料(whole-genome sequencing,WGS),也可以利用轉錄組測序資料(RNA-seq),或者二種技術結合起來更好。
全基因組測序鑒定出的基因融合,基本能确定是由于基因組層面發生某種變異而引起的,但如果沒有轉錄組測序資料,就無法準确判斷融合後産生的新基因是否能夠表達,或表達量的高低。
而轉錄組測序資料鑒定出的基因融合,可以明确是能表達的基因融合,但卻無法完全确定是否是由基因組變異産生的,還是來源于兩個不同基因轉錄後發生的RNA融合。
是以,如果條件允許,同時結合全基因組測序和轉錄組測序來鑒定基因融合,能夠獲得更準确的鑒定結果。
【基因融合鑒定中的常見術語】
在了解基因融合鑒定的方法或軟體前,我們先了解一下基于測序資料鑒定基因融合時的一些常見術語。具體如下圖所示:
基因融合鑒定的一些常見術語(圖檔來源:Liu et al. Nucleic Acids Research, 2016)(A) Intact exon (IE) type andbroken exon (BE) type fusion transcripts; (B) spanning read, split readand anchor length; (C) short and long insert size of DNA fragment forsequencing.
這些常見的術語有:
1)Intact exon (IE) type fusion,是指融合後完整的保留了原來的外顯子,未影響原來的外顯子結構。如上圖A中Gene A的Exon2和Gene B的Exon1融合後完整的保留了兩個外顯子的序列;
2)Broken exon (BE) type fusion,是指融合後沒有保留原來完整的外顯子序列。如上圖A中 Gene A的Exon3的部分序列和Gene B的Exon2融合在一起,融合後的新基因中,來自Gene A的Exon3丢失了部分序列;
3)Breakpoint,是指兩個融合基因在基因組上發生融合的位置,如上圖B中Gene A(藍色)和Gene B(綠色)融合的位點;
4)Spanning read,是指跨越融合位點分别比對到兩個融合基因的paired-end read,比如上圖B中的比對到Gene A(藍色)和Gene B(綠色)的一對read;
5)Split read,是指恰好比對到融合位點上的read,具體如上圖B中右側圖所示;
6)Anchor length,是指跨越融合位點的read左端和右端的長度,具體如上圖B中右側圖所示;
7)short insert size,一般是指雙端測序paired-end sequencing中,兩個read中間間隔的較短距離,一般為幾百bp;
8)long insert size,一般是指雙端測序mate-pair sequencing中,兩個read中間間隔的較長距離,一般為幾kb甚至更長;
基因融合鑒定軟體的開發,一般就是基于上面提到的這些術語,采用相應的算法來設計的。
【基因融合鑒定軟體的性能比較】
到目前為止,已經有數十種不同的基因融合檢測軟體相繼被開發出來,其中一些具有比較好的綜合性能。接下來,我們就進一步比較分析一下一些常用的基因融合鑒定軟體的性能。
下表列出了15種常用的基因融合鑒定軟體在3組不同類型的人工合成資料和3組真實資料集上的性能比較。這些軟體分别是:SOAPfuse,FusionCatcher,JAFFA,EricScript,chimerascan,PRADA,deFuse,FusionMap,TopHat-Fusion,MapSplice,BreakFusion,SnowShoes-FTD,FusionQ,FusionHunter,ShortFuse。
15種基因融合鑒定軟體在3組合成資料和3組真實資料上的F-measure得分比較(圖檔來源:Liu et al. Nucleic Acids Research, 2016)。F-measure是一種統計量,又稱為F-Score,是Precision和Recall權重調和平均,常用于評價分類模型的好壞,數值越高代表性能越好 注:* 綜合表現最好。
值得注意的是,測序read的長度,還有雙端測序read的間隔距離insert size等都會影響基因融合的鑒定效果。是以,上面表中使用了不同類型的測資料來綜合檢驗這15款軟體的性能。其中,Type-1A 為使用wgsim軟體人工合成 的來自5’ 和 3’ end 的chimerical transcripts 雙端測序資料, 其中read長度為100bp,insert size 為500 ± 50 bp; Type-1B資料合成方法和Type-1A類似,隻是雙端測序資料的insert size較小,為 250 ± 50 bp;Type-3B: read 長度為50 bp 的合成資料; 另外三組真實資料分别來自于Breast cancer,Melanoma和Prostate cancer。
比較結果顯示,SOAPfuse,FusionCatcher和JAFFA在3組模拟資料和3組真實資料上的綜合性能最好,取得了最高的F-measure分值。
Liu et al. 進一步比較了15種基因融合檢測軟體在更多真實資料上的性能表現。具體如下圖所示:
15種基因融合檢測軟體在真實測序資料集上的進一步性能比較(圖檔來源: Liu et al. Nucleic Acids Research, 2016 )。A到C縱軸為檢測到的真實基因融合數量,橫軸為15種不同的軟體。D圖為Precision和Recall的曲線比較。其中(A) 和(D)使用的是 Breast cancer資料集; (B) 和 (E) 使用的是Melanoma資料集; (C) 和(F)使用的是Prostate cancer資料集。
真實資料上的比較結果也顯示,SOAPfuse,FusionCatcher和JAFFA在檢測基因融合時具有較高的準确性。
Liu et al. 也比較了15種基因融合檢測軟體在不同測序深度的合成資料集和真實資料集上的運作時間比較。具體如下圖所示:
15種基因融合檢測軟體的運作速度比較(圖檔來源: Liu et al. Nucleic Acids Research, 2016 )。Y軸為運作時間分鐘(min)。A為 合成資料集,read長度為100 bp,模拟測序深度分别為50X, 100X 和200X。B為真實的prostate cancer 171T 的資料集。
運作時間比較結果顯示,FusionMap等軟體的運作速度最快。但前面的結果顯示,FusionMap檢測的基因融合準确性較低。
是以,沒有哪一個方法在各方面的性能比較上面都具有最明顯的優勢。整體來看,SOAPfuse綜合比較最好,接着是FusionCatcher和JAFFA。 而且,因為不同軟體具有的優缺點不一樣,如果聯合多個不同軟體一起鑒定基因融合,或可取得更準确的結果。
釋出者:全棧程式員棧長,轉載請注明出處:https://javaforall.cn/166940.html原文連結:https://javaforall.cn