下面是學員的( 資料挖掘 )直播配套筆記
Oncogenic lncRNA downregulates cancer cell antigen presentation and intrinsic tumor suppression不過不需要看文章,大家隻需要做差異分析即可,這個時候需要注意的是,作者提供的是RPKM值表達矩陣!
1.下載下傳資料GSE113143并加載資料
a=read.table('GSE113143_Normal_Tumor_Expression.tab.gz',sep='\t',quote = "",fill = T,
comment.char = "!",header = T) # 提取表達矩陣
rownames(a)=a[,1]
a <- a[,-1]
複制
2.将FPKM轉換為TPM
Q:為什麼将FPKM轉換為TPM?A:隻有轉換成TPM才勉強可以用limma做差異分析;而DESeq2和edgeR是對count資料進行差異分析
expMatrix <- a
fpkmToTpm <- function(fpkm)
{
exp(log(fpkm) - log(sum(fpkm)) + log(1e6))
}
tpms <- apply(expMatrix,2,fpkmToTpm)
tpms[1:3,]
colSums(tpms)
#輸出結果:
> tpms[1:3,]
N1 N2 N3 T1 T2 T3
0610005C13Rik 0.232 0.1715 0.00 0.00 0.00 0.00
0610007P14Rik 48.391 39.2632 46.04 50.04 59.05 67.29
0610009B22Rik 47.491 58.5954 54.27 49.79 53.13 58.00
> colSums(tpms)
N1 N2 N3 T1 T2 T3
1e+06 1e+06 1e+06 1e+06 1e+06 1e+06
複制
3.差異分析
group_list=c(rep('Normal',3),rep('Tumor',3))
## 強制限定順序
group_list <- factor(group_list,levels = c("Normal","Tumor"),ordered = F)
#表達矩陣資料校正
exprSet <- tpms
boxplot(exprSet,outline=FALSE, notch=T,col=group_list, las=2)
library(limma)
exprSet=normalizeBetweenArrays(exprSet)
boxplot(exprSet,outline=FALSE, notch=T,col=group_list, las=2)
#判斷資料是否需要轉換
exprSet <- log2(exprSet+1)
#差異分析:
dat <- exprSet
design=model.matrix(~factor( group_list ))
fit=lmFit(dat,design)
fit=eBayes(fit)
options(digits = 4)
topTable(fit,coef=2,adjust='BH')
bp=function(g){
library(ggpubr)
df=data.frame(gene=g,stage=group_list)
p <- ggboxplot(df, x = "stage", y = "gene",
color = "stage", palette = "jco",
add = "jitter")
# Add p-value
p + stat_compare_means()
}
deg=topTable(fit,coef=2,adjust='BH',number = Inf)
head(deg)
#save(deg,file = 'deg.Rdata')
複制
這裡面重點就是:RPKM矩陣可以轉為TPM後,再使用limma進行差異分析哦!
4.做完差異分析
## 不同的門檻值,篩選到的差異基因數量就不一樣,後面的超幾何分布檢驗結果就大相徑庭。
if(T){
logFC_t=1.5
deg$g=ifelse(deg$P.Value>0.05,'stable',
ifelse( deg$logFC > logFC_t,'UP',
ifelse( deg$logFC < -logFC_t,'DOWN','stable') )
)
table(deg$g)
head(deg)
deg$symbol=rownames(deg)
library(ggplot2)
library(clusterProfiler)
library(org.Mm.eg.db)
df <- bitr(unique(deg$symbol), fromType = "SYMBOL",
toType = c( "ENTREZID"),
OrgDb = org.Mm.eg.db)
head(df)
DEG=deg
head(DEG)
DEG=merge(DEG,df,by.y='SYMBOL',by.x='symbol')
head(DEG)
save(DEG,file = 'anno_DEG.Rdata')
gene_up= DEG[DEG$g == 'UP','ENTREZID']
gene_down=DEG[DEG$g == 'DOWN','ENTREZID']
}
複制
5.最簡單的超幾何分布檢驗
# 最簡單的超幾何分布檢驗
###這裡就拿KEGG資料庫舉例吧,拿自己判定好的上調基因集進行超幾何分布檢驗,如下
if(T){
gene_down
gene_up
enrichKK <- enrichKEGG(gene = gene_up,
organism = 'mmu',
#universe = gene_all,
pvalueCutoff = 0.05,
qvalueCutoff =0.05)
head(enrichKK)[,1:6]
browseKEGG(enrichKK, 'hsa04512')
dotplot(enrichKK)
ggsave("enrichKK.png")
enrichKK=DOSE::setReadable(enrichKK, OrgDb='org.Mm.eg.db',keyType='ENTREZID')
enrichKK
}
##最基礎的條形圖和點圖
#條帶圖
barplot(enrichKK,showCategory=20)
#氣泡圖
dotplot(enrichKK)
複制

通路與基因之間的關系可視化
#通路與上調基因之間的關系可視化
###制作genlist三部曲:
## 1.擷取基因logFC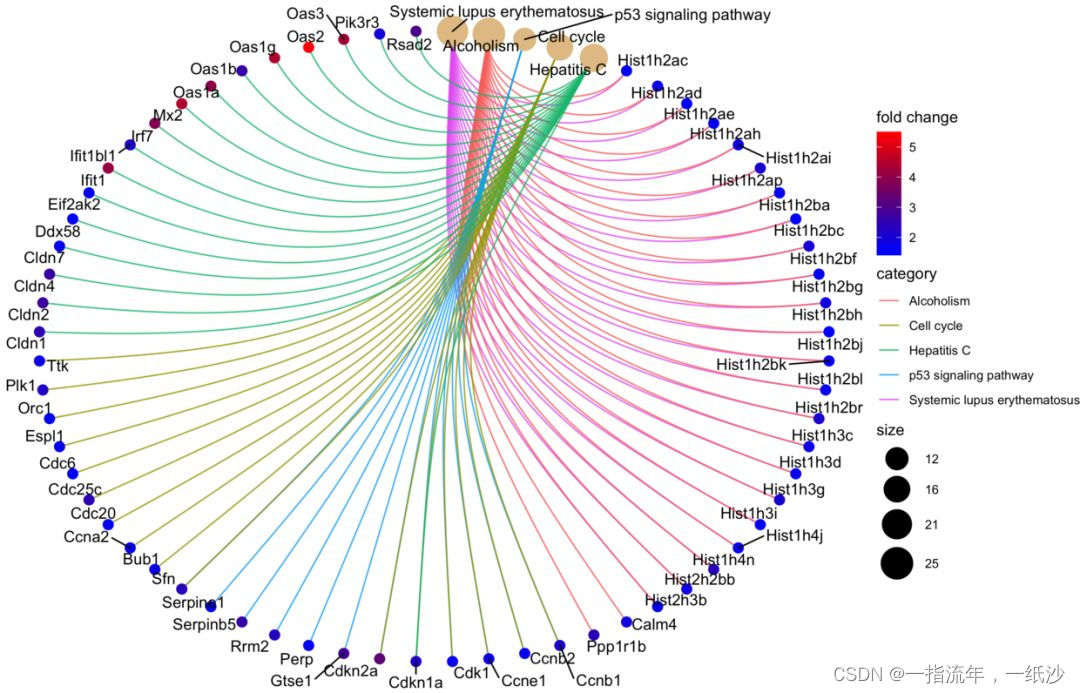
DEG_up <- DEG[DEG$g == 'UP',]
geneList <- DEG_up$logFC
## 2.命名
names(geneList) = DEG_up$ENTREZID
## 3.排序很重要
geneList = sort(geneList, decreasing = TRUE)
head(geneList)
cnetplot(enrichKK, categorySize="pvalue", foldChange=geneList,colorEdge = TRUE)
cnetplot(enrichKK, foldChange=geneList, circular = TRUE, colorEdge = TRUE)
ggsave("enrichKK_cnetplot.png")
複制
通路與通路之間的連接配接展示
#通路與通路之間的連接配接展示
emapplot(enrichKK)
ggsave("enrichKK_emapplot.png")
複制
熱圖展現通路與基因之間的關系
#熱圖展現通路與基因之間的關系
heatplot(enrichKK)
ggsave("enrichKK_heatplot.png")
複制
如果你是做GO資料庫呢,其實還有一個goplot可以試試看
#如果你是做GO資料庫呢,其實還有一個goplot可以試試看
ego_bp_up<-enrichGO(gene = DEG_up$ENTREZID,
OrgDb = org.Mm.eg.db,
keyType = 'ENTREZID',
ont = "BP",
pAdjustMethod = "BH",
pvalueCutoff = 0.01,#0.01
qvalueCutoff = 0.05)
goplot(ego_up)
ggsave("ego_bp_up_goplot.png")
head(ego)
library(stringr)
barplot(ego_bp_up,showCategory = 16,title="The GO_BP enrichment analysis of all DEGs ")+
scale_size(range=c(2, 12))+
scale_x_discrete(labels=function(ego_bp) str_wrap(ego_bp,width = 25))
ggsave("ego_bp_up_barplot.png")
複制