1.seqLogo
seqLogo是1990年發表的一個可視化工具,還是基于grid作圖的工具。可以輸入從MEME,JASPAR等資料庫下載下傳的PPM矩陣(需把header處理掉),即可出圖。
# 安裝
source ( "http://bioconductor.org/biocLite.R" )
biocLite("seqLogo")
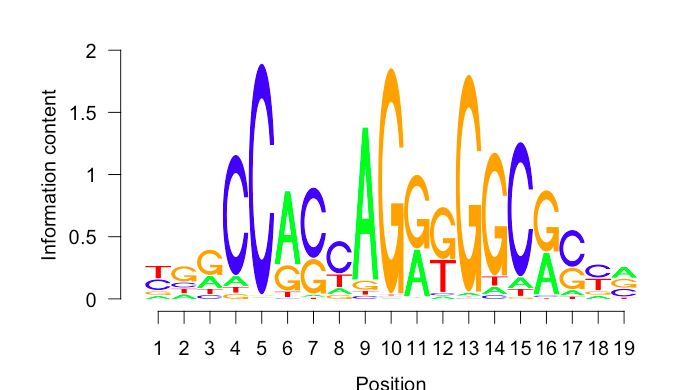
# 導入CTCF的PPM矩陣
library(seqLogo)
ctcf <- read.table('~/Documents/writethings/20200409/ppm.txt', header = F)
# 轉為PWM矩陣(轉置是因為必須要讓行分别是ATCG的PWM score)
trans_ctcf <- makePWM(t(ctcf))
seqLogo(trans_ctcf) 複制
可輸出:
seqLogo
如果想要在一張上輸出多張圖的話(參考https://support.bioconductor.org/p/35240/
):
bad = (sapply( body(seqLogo), "==", "grid.newpage()") |
sapply( body(seqLogo), "==", "par(ask = FALSE)"))
body(seqLogo)[bad] = NULL
tf_names <- c('CTCF', 'SP1')
grid.newpage()
for(i in 0:1){
ppm <- read.table(paste('~/Documents/writethings/20200409/ppm',i+1 ,'.txt', sep = ''), header = F)
pwm <- makePWM(t(ppm))
pushViewport(viewport(x=0.25+0.55*(i%%2),
y=0.5+0.55*(i%/%2),
width=0.5, height=0.6))
seqLogo(pwm)
grid.text(tf_names[i+1], x=0.5, y=1,
hjust=0.5, vjust=1)
popViewport()
} 複制
multi_logo
2.ggseqlogo
這個工具是2017年發在bioinformatics上R包,基于ggplot作圖。可以輸入序列、PFMS和PPM等。
library(ggseqlogo)
ppm <- t(ppm)
rownames(ppm) <- c("A", "G", "C", "T")
ggseqlogo(ppm) 複制
輸出:
ggseqlogo
功能較多,詳細可參考手冊:https://omarwagih.github.io/ggseqlogo/
3.LogoJS
網址:https://logojs.wenglab.org/app/
home
home
可以選擇送出檔案還是粘貼矩陣,檔案中可以包括多個轉錄因子矩陣,輸出:
LogoJS